研究内容 - 微生物機能の解析
1:細菌由来芳香族化合物酸化酵素システムの解析
芳香族化合物の好気的分解では、芳香環に対する二水酸化が最初の反応となることが多く、初発反応という意味で分解系全体の進行を左右することから、分解系の鍵反応と言うことができる。当研究室では、原油中の主要な含窒素芳香族化合物であるカルバゾールに対する初発酸化酵素を材料に、酸化反応メカニズムの解明を行っている。
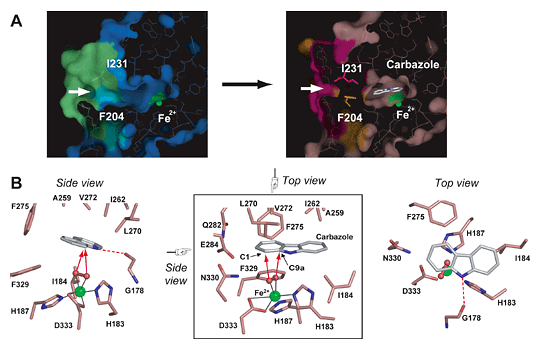
図2-1. Carbazole binding to the substrate-binding pocket of CARDO-O. Panel A illustrates the closure of the entrance of the substrate-binding pocket (white arrows) upon carbazole binding. Conformational changes are obvious for amino acid residues Leu202-Thr214 (shown as cyan [before binding] and orange [after binding]) and Asp229-Val238 (shown as lime [before binding] and magenta [after binding]). Phe204 and Ile231, which had large conformational changes, are shown as stick models. Panel B shows the binding manner of carbazole and oxygen (peroxide ligand) at the substrate-binding pocket (Ashikawa et al. 2006; part of this figure includes unpublished results). The iron ion ligands and amino acid residues that constitute the substrate are shown in stick models and are salmon in color. Carbazole is also shown as a stick model (white for carbon). The iron ions and oxygen (peroxide) ligands are shown as green and small red spheres, respectively. The hydrogen bond interaction between the imino nitrogen of carbazole and the carbonyl oxygen of Gly178 is indicated by a red broken line.
図2-1は、酸化酵素が基質であるカルバゾールを基質結合ポケットに捉えたところをX線結晶構造解析で明らかにしたものである。これにより、カルバゾールの結合後、基質結合ポケットが閉じ基質を逃すことなく酸化する様子が明らかになった。また、基質認識の分子メカニズムが明らかになったことで、基質特異性改変への道筋ができた。
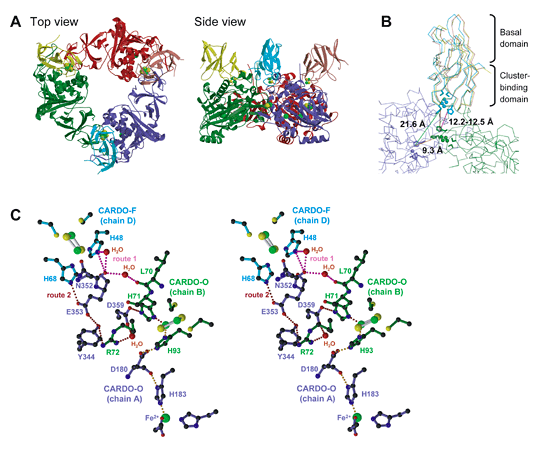
図2-2. (A) Overall structures of the binary complex of CARDO-OJ3 with CARDO-FCA10, (B) superposed view of the three CARDO-FCA10 molecules, and (C) hypothetical electron-transport routes between CARDO-FCA10 and CARDO-OJ3. In panel A, the three CARDO-O subunits (chain A, slate blue; chain B, forest green; and chain C, red) and the three CARDO-F molecules (chain D, cyan; chain E, yellow; and chain F, salmon) are shown. The [2Fe-2S]R and the non-heme iron are shown as spheres (iron ions, green; sulfide ions, yellow). In panel B, the distances between the electron-transfer centers are shown. In panel C, the active-site iron and its coordinated residues of CARDO-O chain A, the [2Fe-2S]R and the coordinated residues of CARDO-O chain B and CARDO-F, and the amino acid residues and water molecules possibly involved in electron transfer in the complex are shown. Each chain is colored as in panel A. The magenta and brown dotted lines indicate the two hypothetical electron-transfer pathways (routes 1 and 2) between the two [2Fe-2S]R. The possible electron-transfer pathway from the [2Fe-2S]R to the active-site iron in CARDO-O is shown as an orange dotted line.
一方、図2-2は酸化酵素と、その電子供与体である3分子のフェレドキシンが、電子授受のための複合体を形成している様子をX線結晶構造解析で明らかにしたものである。当研究室では、さらに、異なる電子供与体と結合する酸化酵素の機能-構造解析を進めており、何が違うと異なる電子供与体を受容できるようになるのかが明らかになりつつある。芳香環酸化酵素は、実際に酸化反応を行う酸化酵素への電子伝達様式の違いから多様なクラスに分類されてきたが、これらの成果は、この分類を、機能-構造相関の観点から見つめ直す手がかりとして高い評価を得ている。
2:難分解性物質分解遺伝子群の分子遺伝学的研究
難分解性物質分解遺伝子群は、細菌ゲノムの中ではプラスミドなどの可動性遺伝因子に含まれている例が非常に多いことが明らかにされている。主としてグラム陰性菌に分布するカルバゾール分解系car遺伝子群も、Pseudomonas属を中心とした細菌では約200kbの環状プラスミドpCAR1に、Sphingomonas属を中心とした細菌では約255kbの環状プラスミドpCAR3に多く見いだされることが明らかにされている(図2-3)。
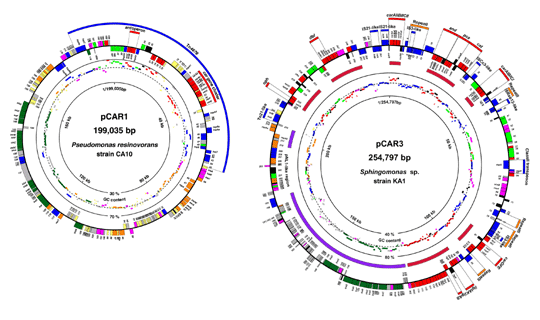
図2-3. The circular gene maps of pCAR1 (left) and pCAR3 (right). Genes or ORFs outside the circle are coded clockwise; those inside are coded counterclockwise. The (putative) functions of genes or ORFs are shown by color as follows: orange, maintenance or DNA processing; dark green, conjugative transfer; blue, transposition or integration; red, degradation; light green, transport; black, regulation; magenta, other known function; gray, unknown function (homologous to hypothetical protein); and yellow, unknown function (no homology). Bars inside the circular gene map indicate the G+C contents of ORFs shown in same color. The broken circle indicates the average G+C content of the entire pCAR1 (56%) and pCAR3 (62.5%). In pCAR3, bold purple lines show the regions homologous to pNL1 (Romine et al. J. Bacteriol., 181, 1585-1602, 1999).
当研究室では、pCAR1やpCAR3の塩基配列決定と、分解遺伝子の転写制御系の解明、プラスミドの基本機能(複製、保持、接合伝達など)の解明を行っている。
3:環境中での汚染物質分解能を制御するプラスミド機能の解明
難分解性物質による汚染を除去するためには、分解力を有する特殊な細菌を取得・培養し汚染サイトに移植するバイオオーグメンテーションが有効であるが、場当たり的に移植するだけでは分解菌が期待する分解力を発揮しない場合が意外に多いのも現状である。分解効果の不確実性の解消には、分解能(分解菌、分解プラスミド)の環境中での振る舞いを分子レベルで詳細に解析し、その理解に立脚して汚染環境に最適化されたバイオオーグメンテーション技術を確立する必要がある(図2-4)。
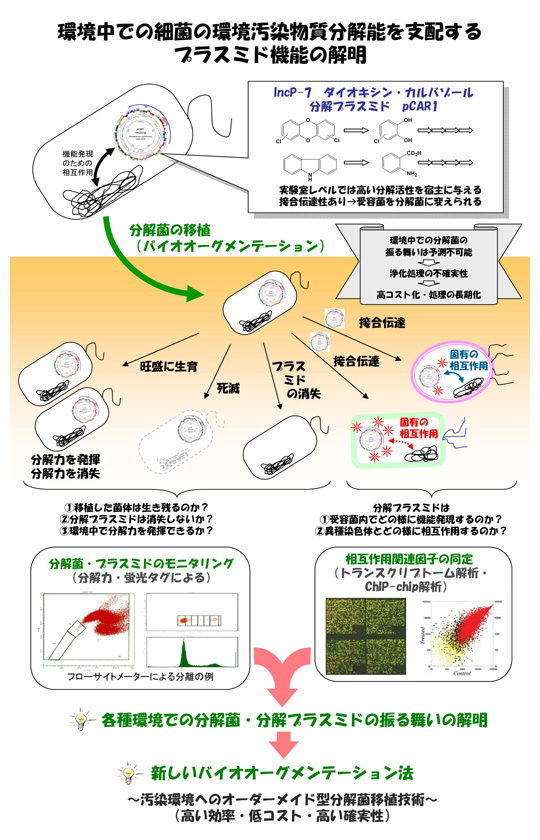
図2-4. Overview of the project entitled ‘Functional Analysis of behavior of Degradative Plasmid in Natural Environment’ [see this page].
そこで、本研究室ではpCAR1とその保持菌が環境中に投与された際の振る舞い(保持度合いや接合伝達頻度など)を分子レベルで解析している。また、これに関連して、接合伝達プラスミドは、異なる宿主内に伝達されると供与菌内とは異なる染色体-プラスミド間転写制御ネットワークを形成し分解酵素遺伝子の発現や保持・複製能などが変化すると考えられるため、このネットワークの多様性・宿主変化による可変性を解明する必要がある。そこで、本研究室ではpCAR1とPseudomonas属細菌を中心とした各種宿主細菌をモデルに、プラスミドと染色体間で形成されるクロストークの実体解明と宿主が変化した場合のクロストークの変化を調べている。
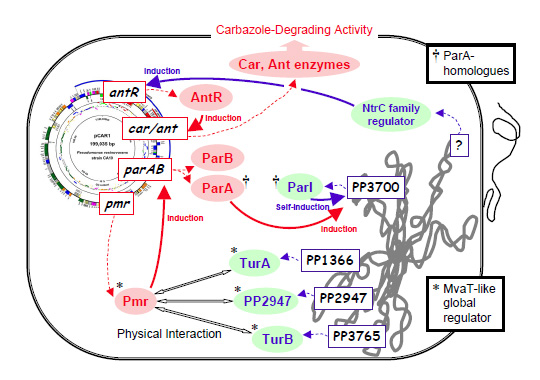
図2-5. Interaction between IncP-7 carbazole degradative plasmid pCAR1 and P.putida KT2440 chromosome detected by transcriptome and biochemical analyses.
図2-5は、マイクロアレイ解析や分子生物学的解析を通して明らかになった、pCAR1とPseudomonas putida染色体間の相互作用の一部を簡単に図示したものである。